《COMPUT STRUCT BIOTEC》丨基于拉曼光谱建立胃癌细胞系分类的集成学习模型
2022年12月,中科院长春光机所李备研究员团队与中山大学肿瘤防治中心赵齐团队应用长光辰英核心产品——PRECI SCS-R300拉曼单细胞分选仪在《Computational and Structural Biotechnology Journal》期刊上发表了题为“Building an ensemble learning model for gastric cancer cell line classification via rapid Raman spectroscopy”的论文。本研究利用拉曼光谱的全光谱、指纹区、高波数区及拉曼背景等光谱数据构建5个光谱数据集,搭建了一个针对不同数据集的stacking集成学习模型——SL-Raman,并实现了胃癌细胞系鉴定。

一、研究背景
细胞错用和交叉污染会影响细胞研究结果的准确性,浪费大量时间、人力和物力。所以细胞系的鉴定十分重要和必要。目前常用的细胞系鉴定方法需要对细胞进行染色或培养。因此有必要开发一种快速、自动的细胞系鉴定新方法。拉曼光谱已经成为微生物鉴定领域的新兴技术之一,具有快速、无创的优点,能够提供生物样品的分子信息,在细胞系鉴定上十分有优势。
二、 研究内容
本研究采用自建的SL-Raman模型对正常和胃癌细胞的拉曼进行分类。我们建立了胃癌细胞系的拉曼光谱数据库,构建了包含不同光谱范围的5个不同的数据集,并充分分析了不同数据集的分类效果。我们正确识别了胃癌细胞,区分了不同分化程度胃癌细胞,还区分了正常与胃癌细胞之间的成分差异。
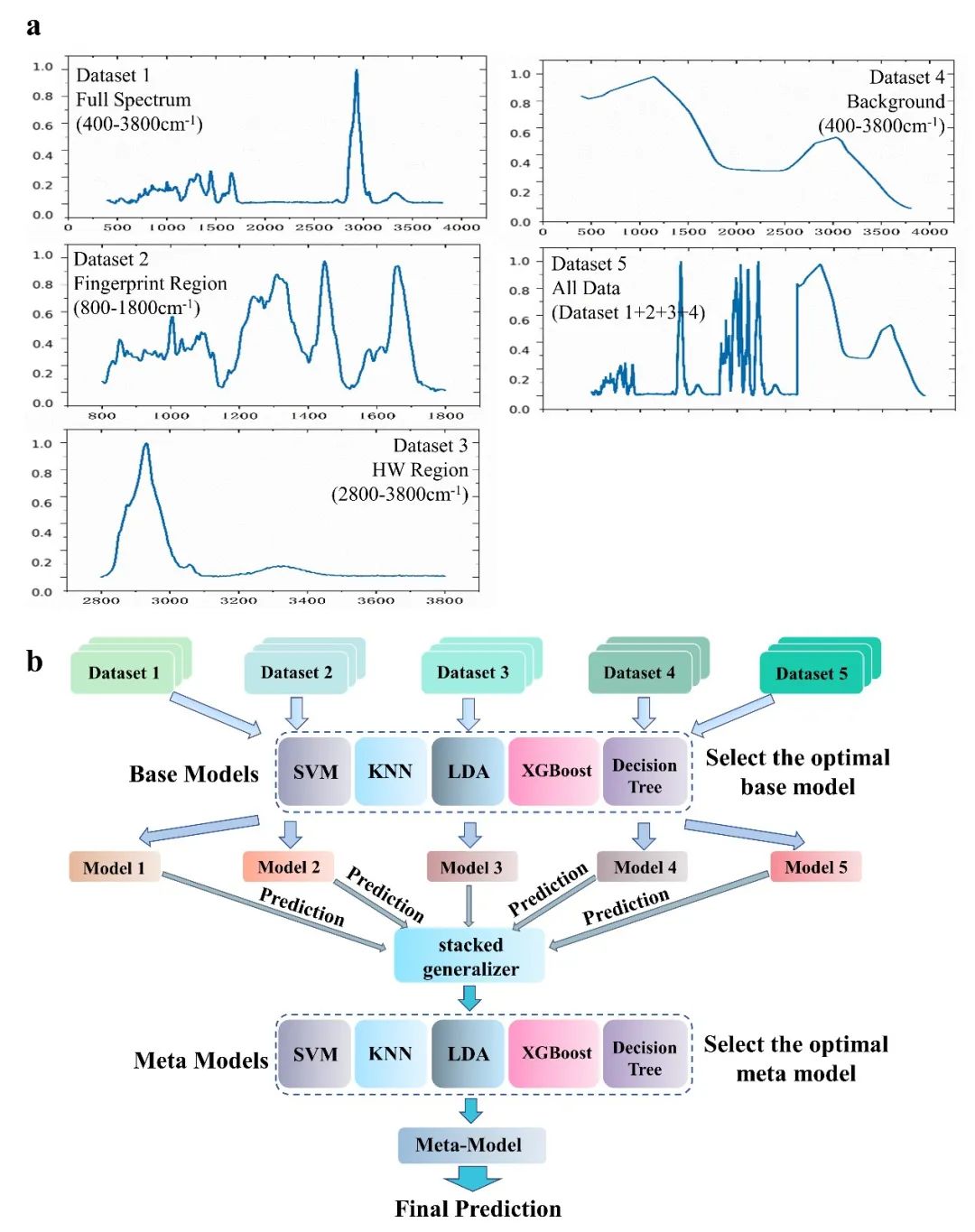
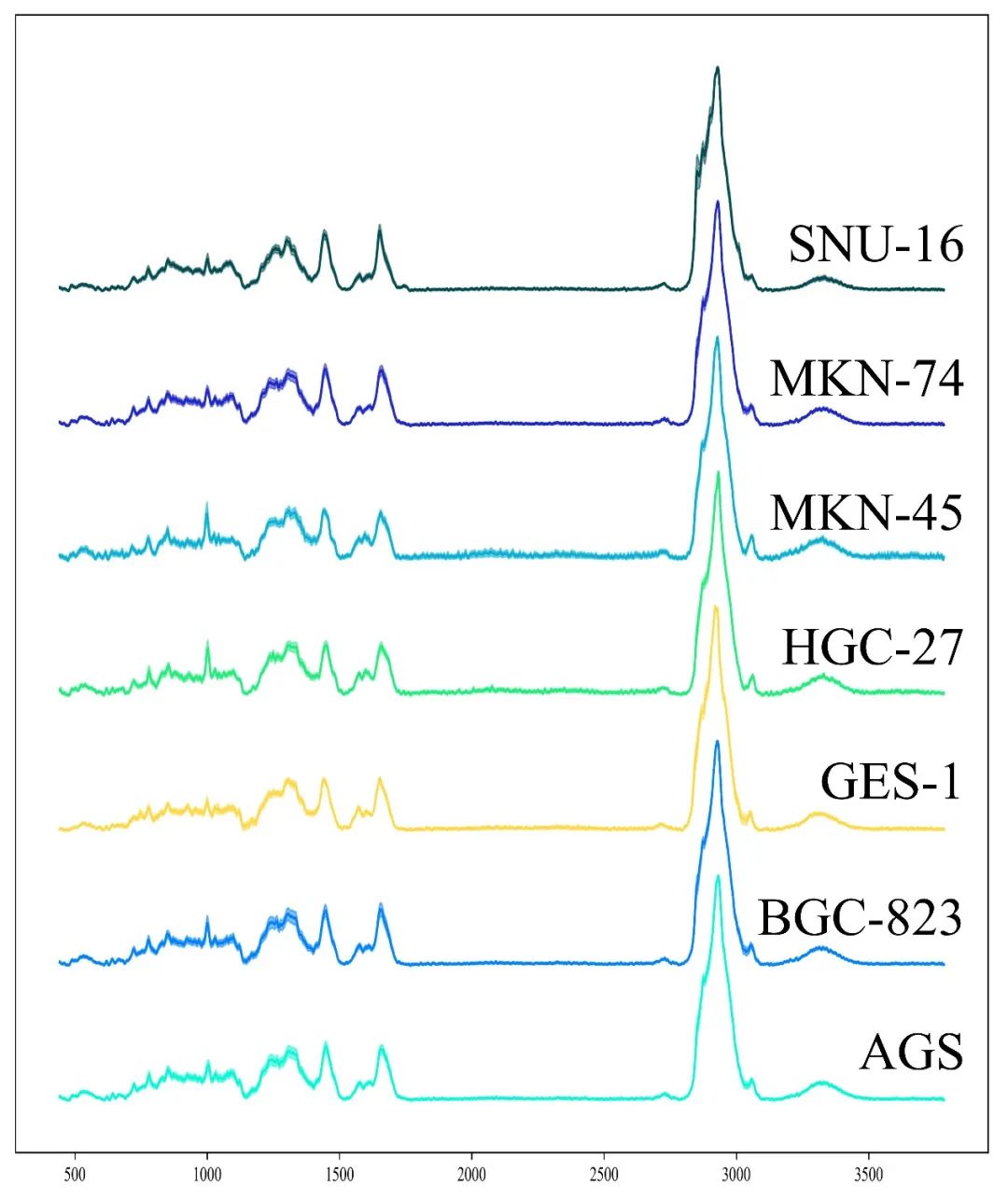
图2. 正常和胃癌细胞的平均拉曼光谱。
(1)区分正常细胞和单个胃癌细胞
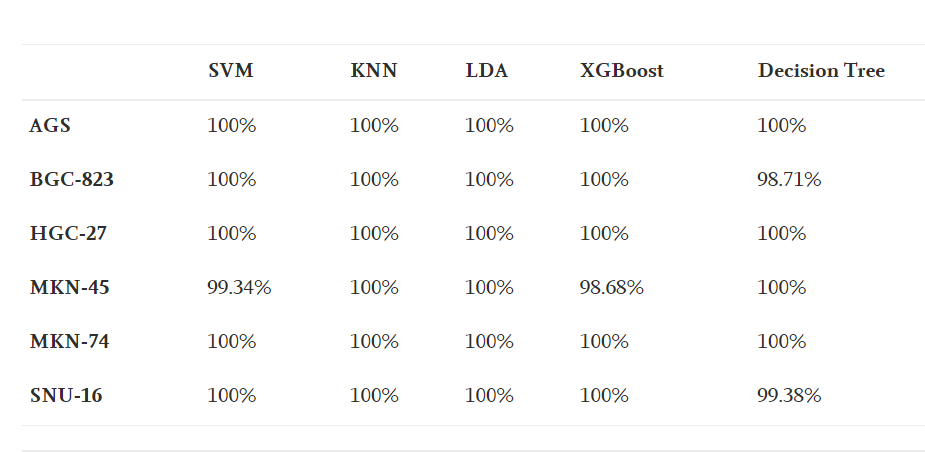
表1. 用SL-Raman鉴定胃癌细胞的准确性。
(2)区分正常细胞与多种类型胃癌细胞
为了验证SL-Raman在不同情况下的分类能力,我们在总共七个正常和胃癌细胞系数据集上训练了该模型。所得到的分类精度、混淆矩阵和接收机工作特性(ROC)曲线如图3所示。当LDA用作SL-Raman的基础模型时,不同元模型的SL-Raman准确度分别为100%、100%、15.15%、100%和99.43%。最终的SL-Raman基础模型是LDA,选择的元模型是KNN,模型精度为100%。
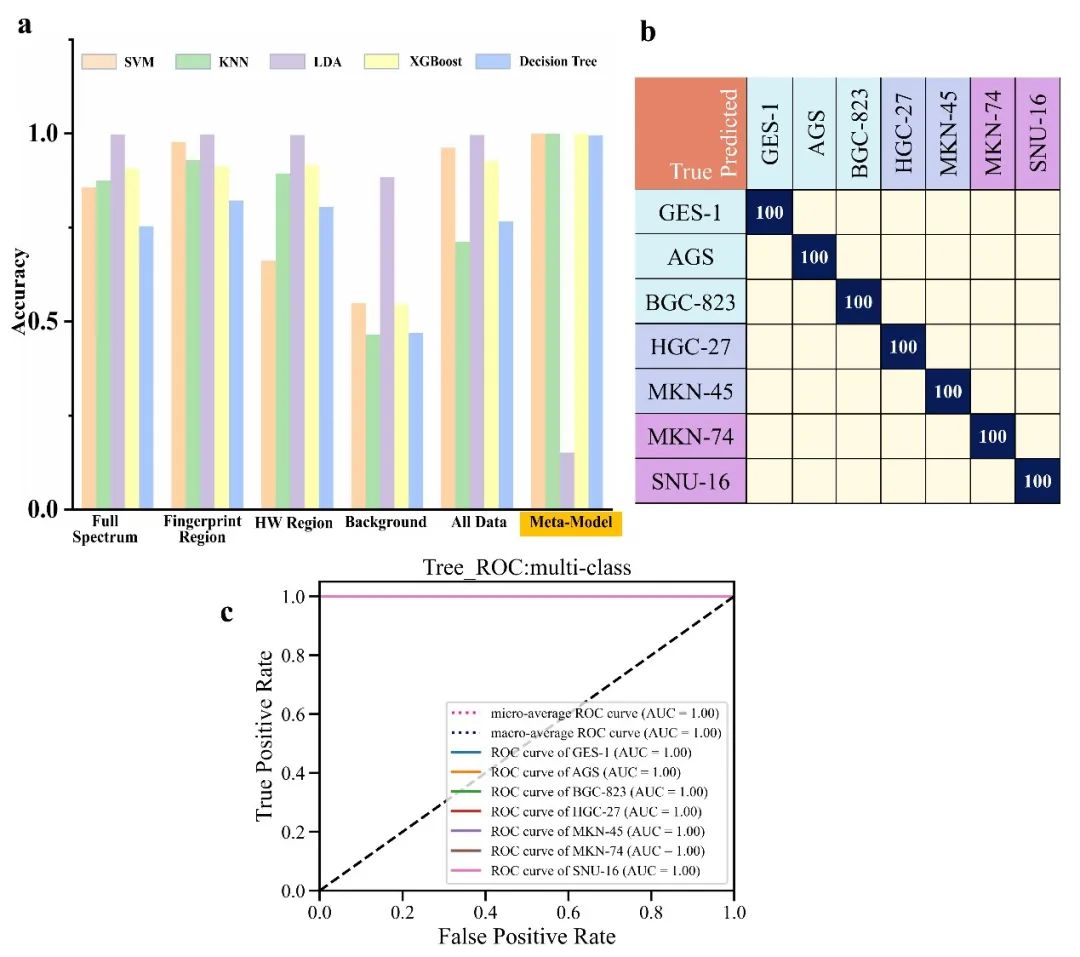
图3. SL-Raman和不同机器学习模型识别7个细胞系的结果。
(3)不同分化程度胃癌细胞的鉴别
为了验证SL-Raman识别不同分化程度胃癌细胞的能力,我们构建了胃癌细胞系AGS(高分化)和BGC-823(低分化)的数据集。分类精度、混淆矩阵和ROC曲线结果如图4所示。如图4所示。当LDA用作SL-Raman的基础模型时,不同元模型的SL-Raman准确度分别为99.36%、99.36%、100%、98.08%和99.36%。最终的SL-Raman基础模型和元模型均为LDA,模型准确度为100%。
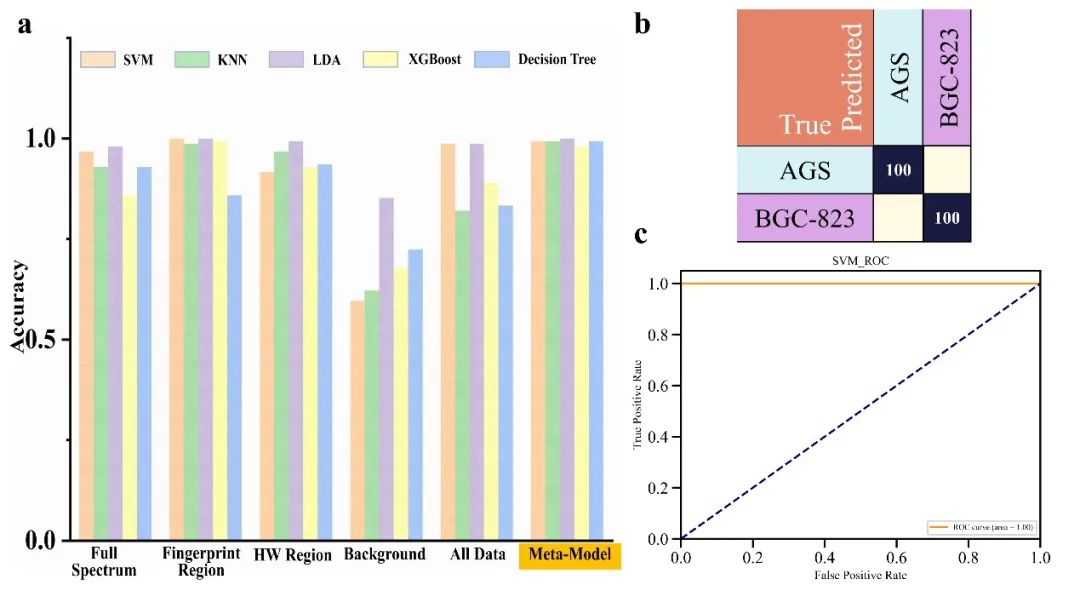
图4. SL-Raman和不同机器学习模型识别不同分化程度
胃癌细胞系的结果。
(4)胃癌细胞的拉曼光谱
拉曼光谱可用于鉴定细胞系和评估细胞的生化成分。如图5.a所示,正常和胃癌细胞的光谱存在显著差异。我们总结了特征峰范围的强度,以直观地比较不同生化成分的总体变化。
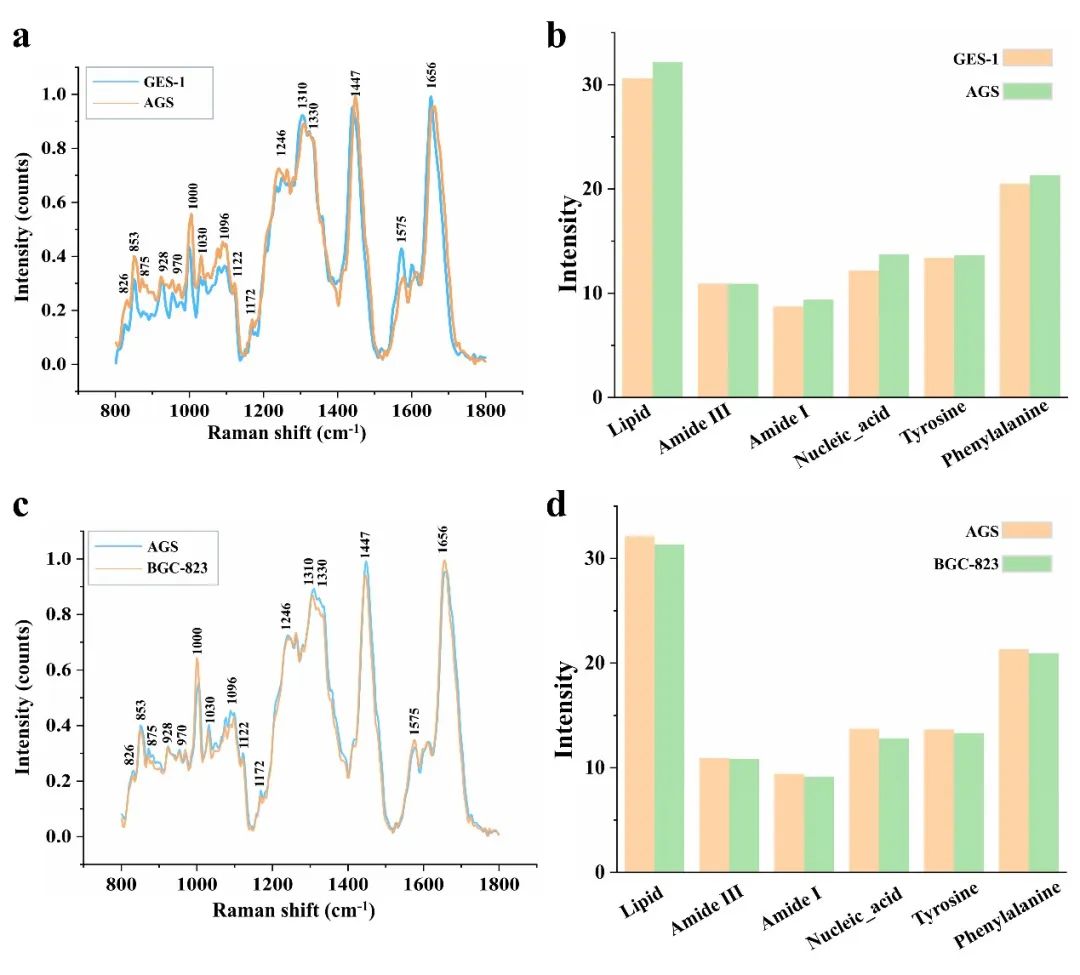
图5.拉曼光谱分析。
三、结论
拉曼光谱可用于识别生物样品中生物成分的差异。拉曼光谱与SL-Raman相结合能够识别正常的胃上皮细胞和胃癌细胞。SL-Raman成功地将各种机器学习技术的优势与多维光谱数据集的数据相结合。该技术提供了一种分析拉曼光谱数据的新方法,因为它可以用少量数据实现高识别精度。
文章链接:
https://doi.org/10.1016/j.csbj.2022.12.050

PRECI SCS-R300拉曼单细胞分选仪







